Introduction
Dans ce document, nous allons implémenter différents algorithmes d’isomorphismes de sous graphes, optimaux ou non.
Dans un premier temps, nous allons nous concentrer sur 3 algorithmes différents, à savoir :
- Weisfeler-Lehman : revenu à la mode avec le noyau sur graphe puis les graph neural network
- Morgan Numbering (TODO): utilisé en chimie, assez similaire à Weisfeler-Lehman dans le principe
- VF2 (TODO): un algorithme d’isomorphisme de graphes reconnu, dévellopé au MIVIA lab de Salerno
Création des graphes
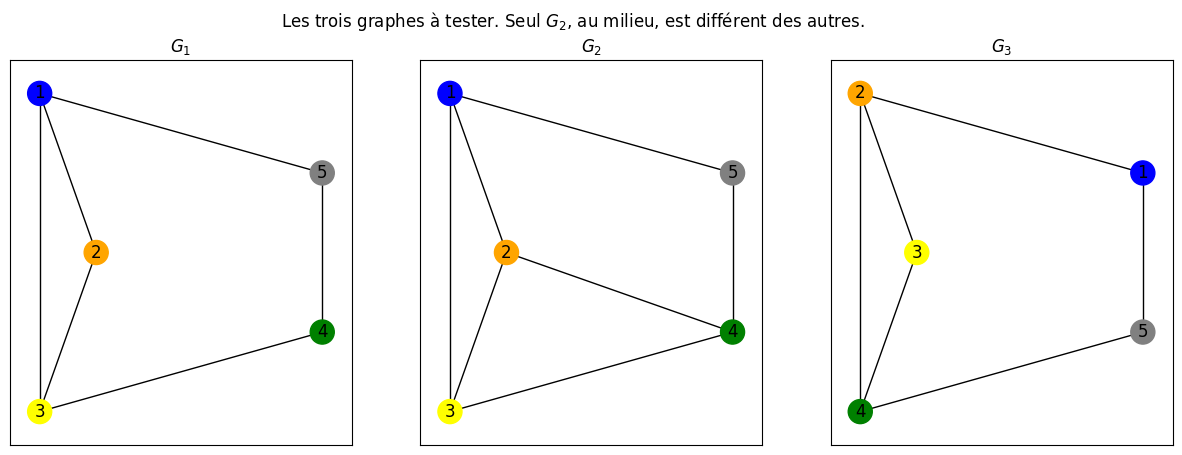
Avant d’implémenter les algos, nous allons créer 3 graphes simples à visualiser. Deux d’entre eux ($G_1$ et $G_3$) sont isormorphes. C’est à dire qu’ils représentent le même graphe, mais que l’ordre des noeuds de chacun des graphes est différent. Il y a une permutation des nœuds.
$G_2$ n’est pas isomorphe à $G_1$ et $G_3$
import networkx as nx
import matplotlib.pyplot as plt
G1 = nx.Graph([[1,2],[2,3],[3,4],[4,5],[5,1],[1,3]])
colors = ["orange","yellow","green","grey","blue"]
pos1 = {1:(0,4), 2:(.2,2),3:(0,0),4:(1,1),5:(1,3)}
for n in G1.nodes():
G1.nodes[n]["label"] = 1
G2 = nx.Graph([[1,2],[2,3],[3,4],[4,5],[5,1],[1,3],[2,4]])
colors = ["orange","yellow","green","grey","blue"]
pos1 = {1:(0,4), 2:(.2,2),3:(0,0),4:(1,1),5:(1,3)}
for n in G2.nodes():
G2.nodes[n]["label"] = 1
# G1 and G3 are isomorphic
G3 = nx.Graph([[1,2],[2,3],[3,4],[4,5],[5,1],[2,4]])
colors = ["blue","orange","yellow","green","grey"]
pos2 = {1:(1,3),2:(0,4), 3:(.2,2),4:(0,0),5:(1,1)}
for n in G3.nodes():
G3.nodes[n]["label"] = 1
fig,axes = plt.subplots(1,3,figsize=(15,5))
nx.draw_networkx(G1,pos=pos1,node_color=colors,ax=axes[0])
nx.draw_networkx(G2,pos=pos1,node_color=colors,ax=axes[1])
nx.draw_networkx(G3,pos=pos2,node_color=colors,ax=axes[2])
for i in range(3):
axes[i].set_title(f"$G_{i+1}$")
fig.suptitle("Les trois graphes à tester. Seul $G_2$, au milieu, est différent des autres.");
Weisfeler - Lehman
Le WL test est un test d’isomorphisme itératif non optimal. C’est à dire que le résultat de l’algorithme n’est pas tout le temps exact au regard de la relation d’isomorphisme entre deux graphes. Si le résultat du test est que les deux graphes sont non isomorphes, alors les deux graphes seront réellement différents. À l’inverse, si le résultat indique que les deux graphes sont isomorphes, le résultat peut être faux pour certains cas particuliers. Ce désavantage est contre balancé par la complexité polynomiale de l’algorithme.
Quelques ressources:
- https://davidbieber.com/post/2019-05-10-weisfeiler-lehman-isomorphism-test/
- https://arxiv.org/pdf/2201.07083.pdf
De manière générale, l’algorithme consiste à calculer une signature du graphe à partir de signatures de chacun des noeuds, chaque signature prenant en compte le voisinage des noeuds. Si l’ensemble des signatures des noeuds des deux graphes sont équivalentes, alors on conclura à un isomorphisme entre les graphes.
Notes
- Chaque noeud se voit attribué un multiset des voisins, implémenté par une liste des voisins ordonnée
- L’ensemble des étiquettes de chaque noeud et leur nombre d’occurences définit la représentation du graphe à comparer
- L’algo s’arrête lorsque les partitions n’ont pas changé entre deux itérations. Deux partitions sont les mêmes si les groupes de noeuds avec un même label sont les mêmes (avec possiblement un label différent entre les deux itérations)
L’algo étape par étape
Caractériser le voisinage
La première étape de l’algo consiste à agréger l’information concernant le voisinage de chacun des noeuds. Par exemple pour le noeud 1 de $G_1$ :
G_test = G1.copy()
labels = dict(G_test.nodes(data="label"))
labels_neighbors = [labels[m] for m in G_test[1]]
labels_neighbors.sort()
print(labels_neighbors)
[1, 1, 1]
Ensuite, les étiquettes des voisins est agrégé avec l’étiquette du noeud courant afin de caractériser la structure locale autour de chaque noeud. Un ‘hash’ est calculé afin d’identifier simplement les environnements similaires.
new_label = [labels[1]] + labels_neighbors
new_desc = hash(tuple(new_label)) # hash sur immutable
G_test.nodes[1]["label"]=new_desc # mise à jour du label
print(G_test.nodes[1]["label"])
print(labels)
-84722638022233667
{1: 1, 2: 1, 3: 1, 4: 1, 5: 1}
Partionnement des nœuds
Une fois l’étape précédente effectuée pour chaque noeud, chaque noeud a un nouveau label caractérisant son voisinage proche. On peut alors calculer une description du graphe en calculant l’histogramme des nouveaux labels. Cela indiquera combien de noeuds ont un voisinage similaire, et cette opération n’est pas sensible aux permutations choisies pour parcourir les noeuds du graphe.
for n in range(2,6): #on met à jour les autres noeuds
print(f"Mise à jour noeud {n}")
labels_neighbors = [labels[m] for m in G_test[n]]
labels_neighbors.sort()
new_label = [labels[n]] + labels_neighbors
print(new_label)
new_desc = hash(tuple(new_label)) # hash sur immutable
G_test.nodes[n]["label"]=new_desc # mise à jour du label
print(G_test.nodes[n]["label"])
Mise à jour noeud 2
[1, 1, 1]
5750192569890809213
Mise à jour noeud 3
[1, 1, 1, 1]
-84722638022233667
Mise à jour noeud 4
[1, 1, 1]
5750192569890809213
Mise à jour noeud 5
[1, 1, 1]
5750192569890809213
def compute_wl_label(graph):
labels = {}
for n in graph.nodes():
label = graph.nodes[n]["label"]
if label in labels:
labels[label].append(n)
else:
labels[label] = [n]
return labels
labels = compute_wl_label(G_test)
print(labels)
{-84722638022233667: [1, 3], 5750192569890809213: [2, 4, 5]}
Ici, on observe que le noeud 2,4 et 5 ont un même label, alors que 1 et 3 ont un label différent. Puisque dans notre cas tous les labels sont égaux, nous avons simplement discriminé sur le degré de chacun des noeuds.
Itérations
Afin de prendre en compte un voisinage de plus en plus grand, les deux étapes précédentes sont répétées jusqu’à que l’une des deux conditions suivantes soient rencontrées :
- les partitions de noeuds n’ont plus bougées : c’est à dire que les mêmes ensemble de noeuds se retrouvent avec un label commun entre deux itérations
- Le nombre d’itérations a atteint la taille du graphe : tout le voisinage a été pris en compte.
#Second itération
labels = dict(G_test.nodes(data="label"))
for n in G_test.nodes(): #on met à jour les autres noeuds
print(f"Mise à jour noeud {n}")
labels_neighbors = [labels[m] for m in G_test[n]]
labels_neighbors.sort()
new_label = [labels[n]] + labels_neighbors
print(new_label)
new_desc = hash(tuple(new_label)) # hash sur immutable
G_test.nodes[n]["label"]=new_desc # mise à jour du label
print(G_test.nodes[n]["label"])
labels = compute_wl_label(G_test)
print(labels)
Mise à jour noeud 1
[-84722638022233667, -84722638022233667, 5750192569890809213, 5750192569890809213]
-7481653653512719100
Mise à jour noeud 2
[5750192569890809213, -84722638022233667, -84722638022233667]
1651756523991284482
Mise à jour noeud 3
[-84722638022233667, -84722638022233667, 5750192569890809213, 5750192569890809213]
-7481653653512719100
Mise à jour noeud 4
[5750192569890809213, -84722638022233667, 5750192569890809213]
-224185658846573216
Mise à jour noeud 5
[5750192569890809213, -84722638022233667, 5750192569890809213]
-224185658846573216
{-7481653653512719100: [1, 3], 1651756523991284482: [2], -224185658846573216: [4, 5]}
Au bout de la deuxième itération, on observe que la partition 2,4,5 a été splitté en deux : 2 et 4,5. Donc on continue !
Conclusion du test d’isomorphisme
Lorsque nous allons appliquer l’algorithme itératif décrit plus haut à chacun des deux graphes à comparer, nous obtenons une partition pour chacun des graphes. La conclusion du test d’isomophisme est déduite de la comparaison des histogrammes de chacune des partitions. Si les deux graphes ont le même nombre de noeuds avec un label similaires, alors nos deux graphes sont potentiellement isomorphes. À l’inverse, si les histogrammes sont différents, alors nous pouvons conclure avec certitude que les deux graphes sont isomorphes.
# Calcul de l'histogramme des labels de G_test
histogramme = {label : len(labels[label]) for label in labels}
print(histogramme)
{-7481653653512719100: 2, 1651756523991284482: 1, -224185658846573216: 2}
Version finale du code
def wl_test_id(graph):
LABEL_WL = 'wl'
"""
Returns a dictionnary labels LABEL_WL as keys and list of nodes having this label as value iterative wl
"""
def compute_wl_label(graph):
labels = {}
for n in graph.nodes():
label = graph.nodes[n][LABEL_WL]
if label in labels:
labels[label].append(n)
else:
labels[label] = [n]
return labels
def compute_wl_signature(graph):
"""
Compute an histogram from the dicts of labels.
"""
labels = compute_wl_label(graph)
return {label : len(labels[label]) for label in labels}
def compute_partition(graph):
"""
Returns a list where each element is a list of nodes having the same label
"""
labels = compute_wl_label(graph)
partition = list(labels.values())
[l.sort() for l in partition]
partition.sort()
return partition
iter = 0
old_partition = {}
conv = False
# Initialisation des labels
for n in graph.nodes():
graph.nodes[n][LABEL_WL] = graph.nodes[n]["label"]
# max number of iterations is the number of nodes
while (iter < graph.order() and not conv):
#compute tuple
old_labels = dict(graph.nodes(data=LABEL_WL))
for n in graph.nodes():
# we compute the new label
labels_neighbors = [old_labels[m] for m in graph[n]]
labels_neighbors.sort()
# compute hash
new_label_list = [old_labels[n]] + labels_neighbors
new_desc = hash(tuple(new_label_list)) # hash sur immutable
graph.nodes[n][LABEL_WL]=new_desc
# partitions
partition = compute_partition(graph)
if partition == old_partition:
conv = True
old_partition=partition
iter += 1
return compute_wl_signature(graph)
def wl_test(graph_1,graph_2):
return wl_test_id(graph_1) == wl_test_id(graph_2)
print(f"$G_1$ $G_3$ sont isomorphes : {wl_test(G1,G3)}")
print(f"$G_1$ $G_1$ sont isomorphes : {wl_test(G1,G1)}")
print(f"$G_1$ $G_2$ sont isomorphes : {wl_test(G1,G2)}")
$G_1$ $G_3$ sont isomorphes : True
$G_1$ $G_1$ sont isomorphes : True
$G_1$ $G_2$ sont isomorphes : False
Conclusion
Ce test d’isomorphisme, rapide à implémenter et à comprendre a été remis au gout du jour par les noyaux sur graphes (Weisfeler Lehman Graph kernel) ainsi que par l’étude théorique de la capacité des Graph Neural Networks à distinguer deux graphes différents.